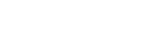机器之心报道
编辑:杜伟、小舟
我们距离精确预测 DNA 结构还远吗?目前 , 使用人工智能预测化合物分子结构是一个火热的研究课题 , DeepMind 蛋白质结构预测工具 AlphaFold2 证明了这一点 。 但应看到 , 实现分子结构准确预测的背后需要庞大的数据集 。 斯坦福大学的一项研究打破了这一限制 , 他们提出的机器学习方法仅使用很少的数据即实现了准确的 RNA 结构预测 。
确定生物分子的 3D 形状是现代生物学和医学发现中最困难的问题之一 。 许多公司和研究机构花费数百万美元来确定分子结构 , 却也常常无果 。
来自斯坦福大学的研究团队利用机器学习的方法解决了这个难题 。 在计算机科学系副教授 Ron Dror 的指导下 , 斯坦福大学博士生 Stephan Eismann 和 Raphael Townshend 巧妙地使用机器学习技术开发了一种通过计算预测生物分子准确结构的方法 。 并且即使仅从少数已知结构中学习 , 他们的方法也能成功 , 使其适用于结构最难通过实验确定的分子类型 。
8 月 27 日 , 该团队与斯坦福大学生物化学系副教授 Rhiju Das 合作的研究论文在《Science》上发表并登上封面 。

文章图片
论文地址:http://science.sciencemag.org/content/373/6558/1047
在此之前 , 去年 12 月该团队的一篇研究论文已经登上了生物医学期刊《Proteins》 。

文章图片
论文地址:https://onlinelibrary.wiley.com/doi/10.1002/prot.26033
在《Proteins》的论文中 , 研究团队介绍说:该研究建立的神经网络架构从包含数万个原子的分子结构中进行端到端的学习 , 其中涉及基于点的原子表示、旋转和平移的等变性、局部卷积和分层子采样操作 。
两篇论文的主要作者 Townshend 说:「结构生物学是对分子形状的研究 , 结构决定功能 。 」该团队设计的算法不仅可以预测准确的分子结构 , 还能够解释不同分子的工作原理 , 该方法将适用于基础生物学研究、药物研发等 。 具体来讲 , 团队成员 Eismann 以蛋白质举例说明:「蛋白质是执行各种功能的分子机器 。 为了执行它们的功能 , 蛋白质通常会与其他蛋白质结合 。 如果已知一对蛋白质与疾病有关 , 并且知道它们在三维条件下如何相互作用 , 医学上就可以尝试用一种药物非常具体地针对这种相互作用 。 」
该研究的方法已经在蛋白质复合物和 RNA 分子方面取得了成功 。 正如研究团队成员 Dror 所说:「机器学习近来取得的大多数进展都需要大量数据进行训练 。 而该研究的方法在训练数据很少的情况下取得成功的事实意味着:相关方法可以解决许多数据稀缺的领域中未解决的问题」 , 因此该方法可能具有巨大潜力 。
使用少量数据实现 RNA 准确结构预测
RNA 分子的 3D 结构对 RNA 分子发挥自身功能至关重要 , 在药物发现中也很有意义 。 然而 , 已知的 RNA 结构很少 , 并且通过计算来预测 RNA 结构极具挑战性 。
而斯坦福大学的这项研究使用机器学习(ML)的方法 , 只使用 18 种已知的 RNA 结构进行训练 , 就能够识别出准确的结构模型 , 同时无需这些结构模型的定义特性 。 通过这种机器学习方法得到的评分函数——原子旋转等变评分器(Atomic Rotationally Equivariant Scorer, ARES)显著优于以往方法 。
下图为训练集中的 18 种 RNA 结构图示 。

文章图片
具体地 , 为了训练 ARES , 研究者使用了 1994 年至 2006 年之间已发表的 18 个 RNA 分子 , 并利用 Rosetta FARFAR2 采样方法生成了每个 RNA 的 1000 个结构模型 , 同时没有使用任何已知结构 。 接着 , 他们优化了 ARES 神经网络的参数 , 使其输出尽可能匹配每个模型对应结构的均方根误差(RMSD) 。
下图 1 为 ARES 网络 , 其中 A 表示:仅给出原子坐标和元素类型 , ARES 即可预测结构模型的准确率;B 表示利用 ARES 的 RNA 结构预测;C 表示包含 18 种已有小型 RNA 结构的训练集;D 表示包含新的、更大 RNA 结构的基准集 。
文章图片
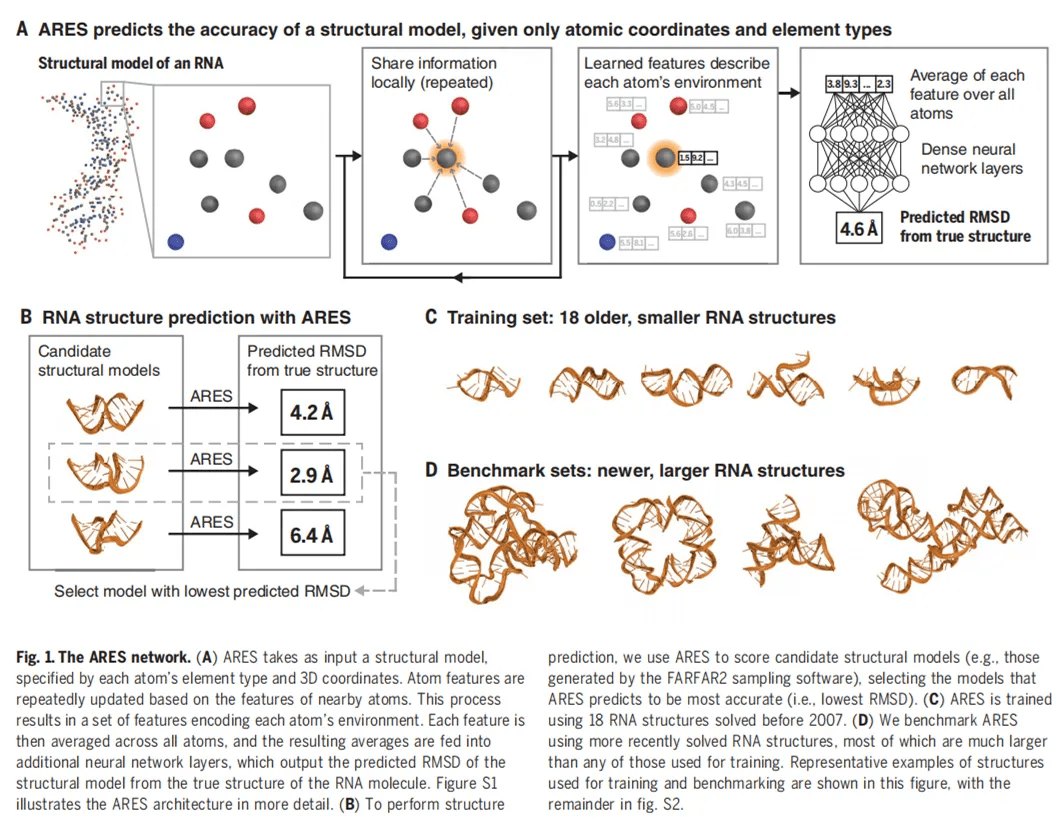
下图 2 展示了 ARES 显著优于以往识别准确结构模型的评分函数 。 A 表示评分最佳结构模型的 RMSD;B 表示 10 个评分最佳结构模型中最低的 RMSD;C 表示无限接近评分最佳结构模型的排名;D 表示 2007 年以来评分最佳结构模型的 RMSD 。
文章图片
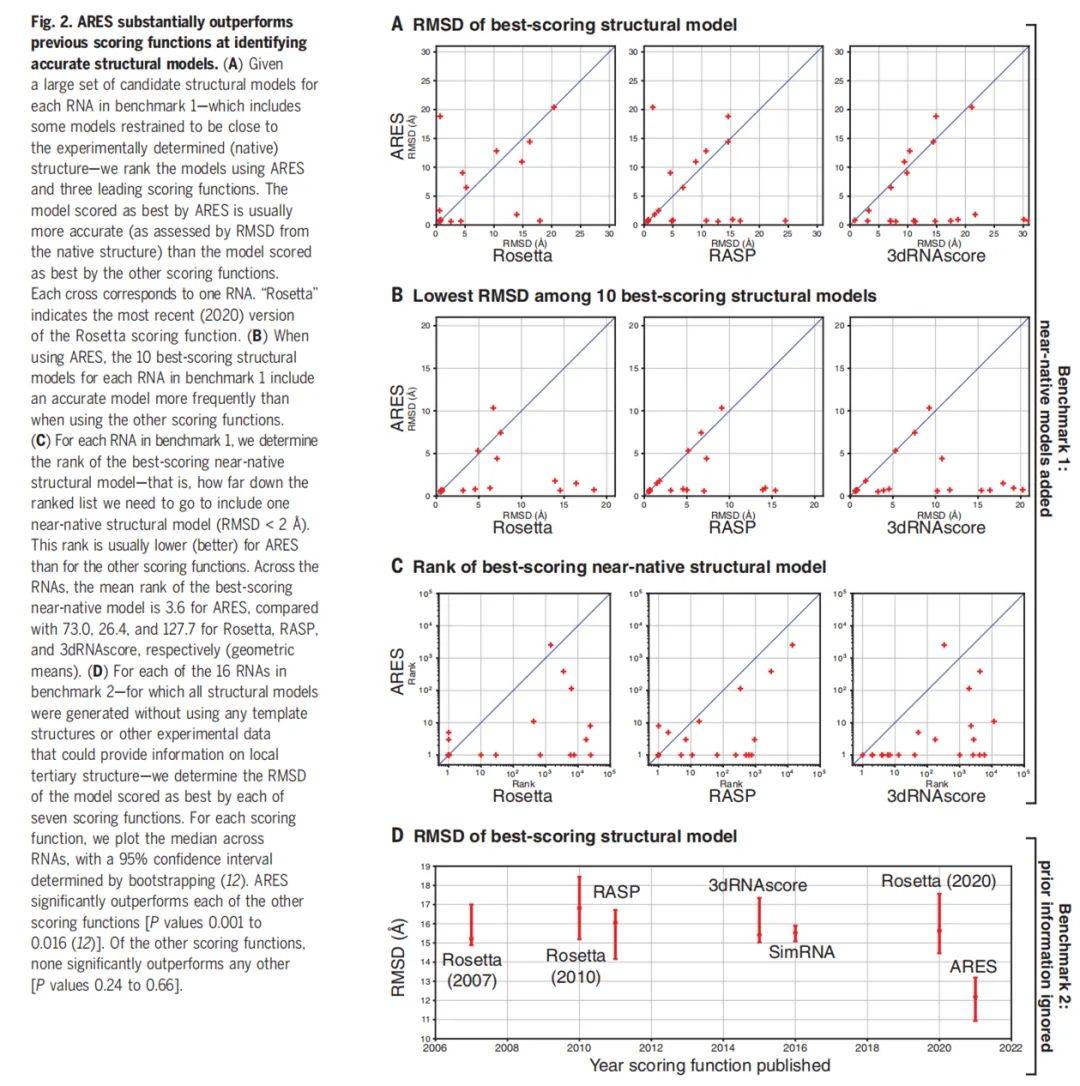
ARES 在社区范围内的 Blind RNA 结构预测挑战赛「RNA Puzzles」中实现了 SOTA 结果 。 如下图 3 所示 , A 表示 ARES 与其他方法的 Blind 预测准确率结果对比;B 表示 ARES 预测的晶体结构;C 表示其他方法实现的最佳晶体结构预测 。
文章图片
此外 , ARES 还能学习识别事先没有明确说明的 RNA 结构的关键特征 。 下图 4A 表示 ARES 学习螺旋宽度以实现最优的碱基配对;4B 表示学习识别关键的 RNA 特征 。
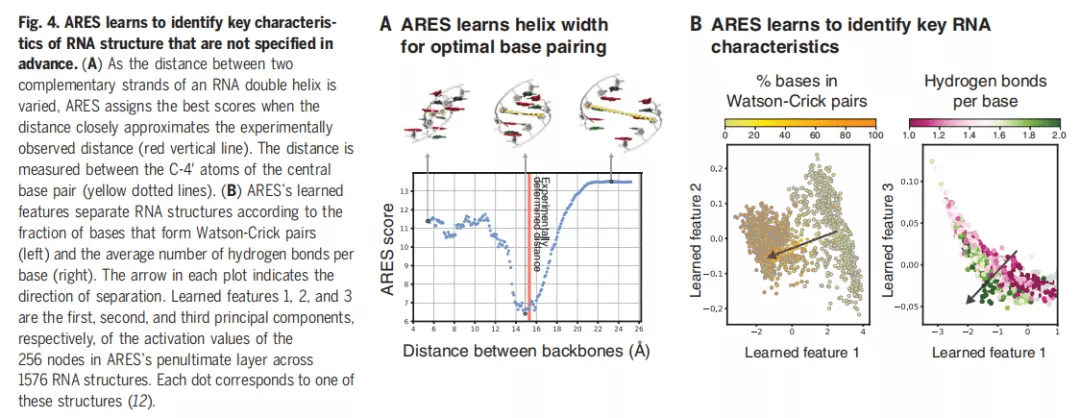
文章图片
就其优势而言 , 斯坦福大学的方法可以基于少量数据进行有效学习 , 克服了标准深度神经网络的主要限制 。 并且该方法仅使用原子坐标作为输入 , 不包含特定的 RNA 信息 , 因此适用于解决结构生物学、化学、材料科学等领域的各种问题 。
作者简介

文章图片
共同一作 Raphael Townshend 是分子和药物设计 AI 初创公司 Atomic AI 的 CEO 。 目前 , 他在斯坦福 AI 实验室攻读博士 , 研究兴趣包括机器学习、结构生物学、高性能计算和计算机视觉 。

文章图片
通讯作者之一 Ron Dror 是斯坦福大学计算机科学系副教授 , 也是斯坦福 AI 实验室所属一个研究小组的负责人 , 专注于使用计算机技术解决生物学和药物发现的基础问题 。 作为高性能计算、机器学习、图像分析、结构生物学和药物设计等领域的专家 , 他还教授计算机生物学和机器学习 , 并为技术和制药企业提供咨询 。
参考链接:
https://raphael.tc.com/
https://drorlab.stanford.edu/rondror.html
https://news.stanford.edu/2021/08/26/ai-algorithm-solves-structural-biology-challenges/
【模型|深度学习精确预测RNA,仅需训练18种已知结构:研究登上Science封面】https://www.genengnews.com/news/rna-structures-predicted-with-uncanny-accuracy-by-unbiased-ai/
推荐阅读
- MateBook|深度解析:华为MateBook X Pro 2022的七大独家创新技术
- 问答|紧追B站加码知识类内容,抖音上线“学习频道”
- Siamese|一个框架统一Siamese自监督学习,清华、商汤提出简洁、有效梯度形式,
- 技术|探秘AI智慧之旅,科大讯飞AI学习机研学游第一期圆满落幕
- 人物|最有深度的8个公众号,你关注了吗
- 模型|2022前展望大模型的未来,周志华、唐杰、杨红霞这些大咖怎么看?
- 材料|别在玩吃鸡,小学生都开始学习3d打印技术了
- 字节跳动|抖音上线学习频道,为知识内容增加一级入口
- 数据|抖音上线学习频道,位于一级入口
- 国际|特奖得主任队长,清华夺冠NeurIPS 2021国际深度元学习挑战赛