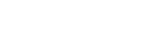8月2日 , 西北大学孙士生教授课题组与西安电子科技大学张军英教授课题组联合团队的重要研究成果在线发表在Nature Methods杂志上 。 成果以“StrucGP: de novo structural sequencing of site-specific N-glycan on glycoproteins using a modularization strategy”为题 , 介绍了一种可解析糖蛋白上糖链序列的新方法及其配套软件StrucGP , 并描述了其技术细节 。

文章图片
图/在线发表
糖类和蛋白质均是四大生物大分子之一 , 在生命活动中发挥非常重要的作用 。 其中N-连接糖蛋白主要存在于细胞膜表面和各种体液中 , 参与细胞间识别和通讯、免疫应答、病原体的宿主识别等重要生物学过程 。 很多糖蛋白已经成为重要的疾病生物标志物和药物靶标 。 目前 , 同时解析糖基化位点和糖链组成信息已基本可以实现 , 但由于糖链结构的复杂性和糖基化微不均一性特性 , 糖基化位点特异性的糖链序列测定(也称糖链结构解析)依然存在极大挑战 。
文章图片
图/StrucGP原理图
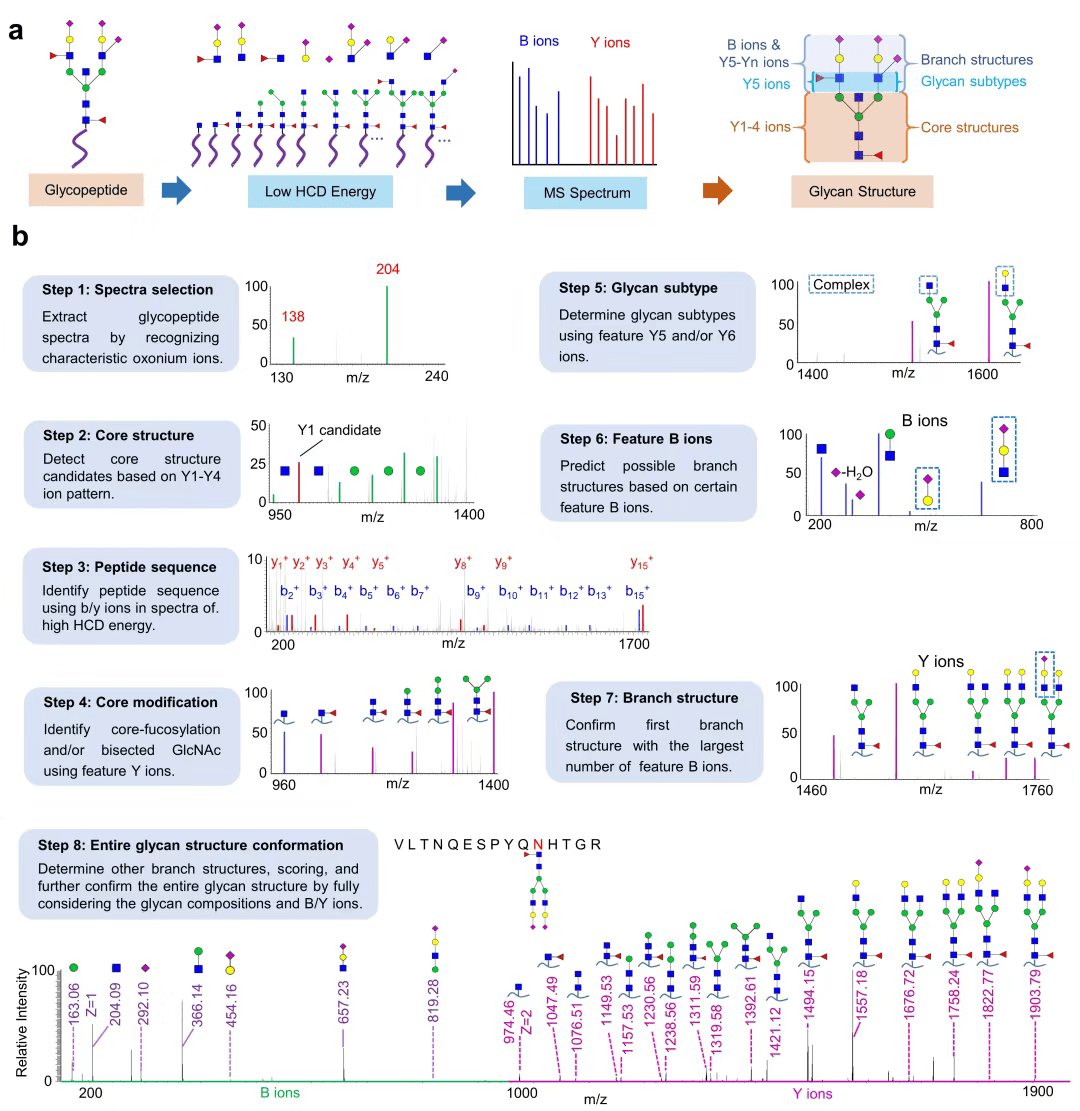
孙士生教授团队建立的新方法中 , StrucGP采用了模块化的解析策略 , 首先将N-连接糖链结构分为核心结构、糖链类型以及分支结构三个部分 , 分别进行从头结构解析 , 然后合并获得糖链的整体结构信息 , 极大地降低了糖链结构解析的复杂度 , 提高了糖链结构解析的准确性 。 另外 , StrucGP在糖链结构解析中并不依赖于糖链数据库 , 因此有助于新的糖链结构鉴定 。 在鉴定准确度方面 , 研究团队建立了基于诱饵数据库(decoy database)和诱饵谱图 (decoy spectra) 的假阳性率评估方法 , 以及多种对糖链各模块精细结构的置信度(probability)评估方法 , 有助于研究人员对鉴定结构进行可信度评估 。

文章图片
【位点|西北大学孙士生教授团队建立位点特异性糖链序列测定新方法】 图/孙士生教授团队合影
该技术首次实现了完整糖肽上N-连接糖链序列的解析 , 为糖蛋白质组学研究提供了新的技术手段 , 在生物医药领域包括新的生物标志物和治疗靶点发现、重要糖蛋白抗体药物的糖基化分析、病毒上重要糖蛋白的糖基化解析、疾病致病机理研究等方面都具有广泛的应用前景 。 比如在肿瘤研究中 , StrucGP能够直接获得肿瘤特异性糖链结构所修饰的糖蛋白和糖基化位点等信息 , 从而获得其亚细胞定位、信号通路、可能的分子功能等关键信息 , 有助于研究者进一步深入研究特定糖链/糖蛋白的生物学功能(实例可参考Jia L. et al. Theranostics, 2021) 。
西北大学生命科学学院博士生申洁晨、贾丽、青年教师党刘毅博士和西安电子科技大学硕士生苏远杰为本论文的共同第一作者 , 孙士生教授为通讯作者 。 孙士生教授团队自2017年创建以来一直从事糖蛋白质组学方法学和应用研究 , 致力于建立可全面和精细解析N-糖蛋白质组的方法体系 , 以及开发可实现高通量和自动化解析完整糖肽的软件系统 。 团队研究成果先后在Nature Biotechnology, Nature Methods, Nature Communications, Theranostics, Analytical Chemistry 等期刊发表 。
该研究得到国家自然科学基金委“生物大分子动态修饰与化学干预”重大研究计划、面上和青年项目 , 科技部重点研发项目和中国博士后基金等多项科研项目的资助 。
全文链接:https://doi.org/10.1038/s41592-021-01209-0
推荐阅读
- 大学|教老年人“玩转”智能手机!太原一老年大学特色课程越来越火
- 文件|日本惠普公司误删京都大学超算系统77TB重要数据
- 误删除|日本惠普公司闯大祸,京都大学超算系统 77TB 重要数据被误删
- 导论|时隔五年,普林斯顿大学经典书《在线凸优化导论》第二版发表
- HP|日本惠普公司误删京都大学超算系统77TB重要数据
- 警告!|一大学生骂外卖员是狗 校方回应称已成立调查组
- IT|牛津大学科学家:omicron造成的疾病跟一年前新冠病毒“不一样”
- 人工智能|正片来了!五期「AI大咖说」第一期,探秘清华大学AI&机器人实验室
- 人物|云南一女大学生辱骂骑手是“送外卖的狗”:校方回应
- 技术|四川大学化学工程教师团队:做磷化工领域的忠诚卫士