蛋白质是生命的物质基础 , 每个蛋白质的氨基酸链扭曲、折叠、缠绕成复杂的结构 , 想要破解这种结构通常需要花很长的时间 , 甚至难以完成 。 截至目前 , 约有10万个蛋白质的结构已经用实验方法得到了解析 , 但这在已经测序的数10亿计的蛋白质中只占了很小一部分 。
在蛋白质结构解析的几十年历史中 , X射线晶体学、核磁共振波谱学(NMR)、冷冻电镜(Cryo-SEM)技术纷纷发挥了巨大的贡献 。 同时 , 通过蛋白质的氨基酸序列来预测其结构这一设想也在50多年前就已提出 。 2020年底 , 谷歌旗下人工智能公司DeepMind的新模型AlphaFold2带来了巨大突破 , 在蛋白质结构预测准确性方面达到接近人类实验结果 , 让整个结构生物学界震惊 。
实际上 , 国内的学术界和产业界也在蛋白质结构预测领域紧追国际步伐 。 澎湃新闻(www.thepaper.cn)采访人员获悉 , 12月8日 , 北京深势科技有限公司(下称“深势科技”)宣布推出蛋白质结构预测工具 Uni-Fold , 这是蛋白质结构研究领域首款完全开源并成功复现大规模训练的工具 。
据介绍 , 在相同的测试条件下 , Uni-Fold 的预测精度超越了华盛顿大学蛋白设计研究所David Baker教授课题组研发的RoseTTAFold , 与DeepMind官方发布的AlphaFold2模型接近 。 深势科技团队使用Uni-Fold对CASP14蛋白预测任务中的序列数据集进行了直接测试 , 平均 Cα-lDDT 达到82.6 。 此外 , Uni-Fold 的推理代码更加轻量、高效 , 在相同硬件环境下 , 能够获得与公布的AlphaFold2代码相比2-3倍的效率提升 。
文章图片
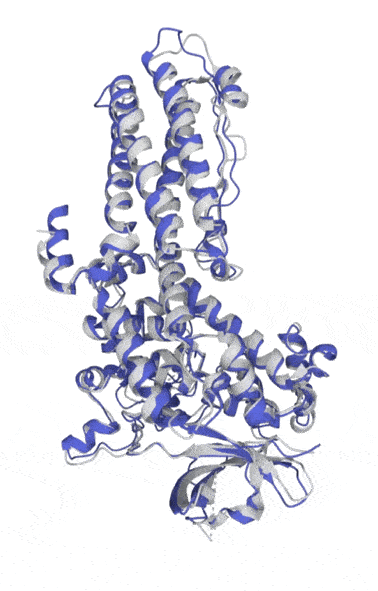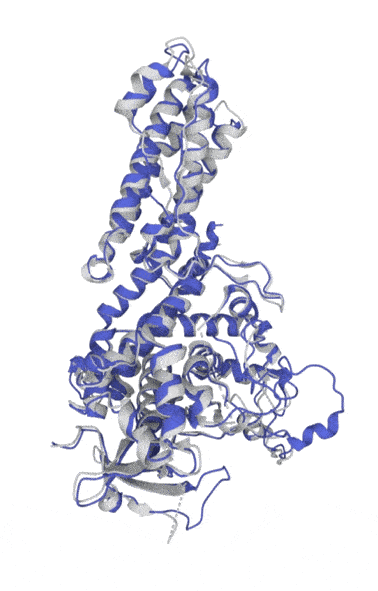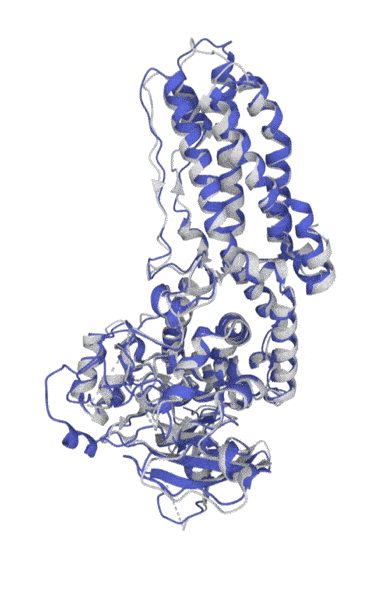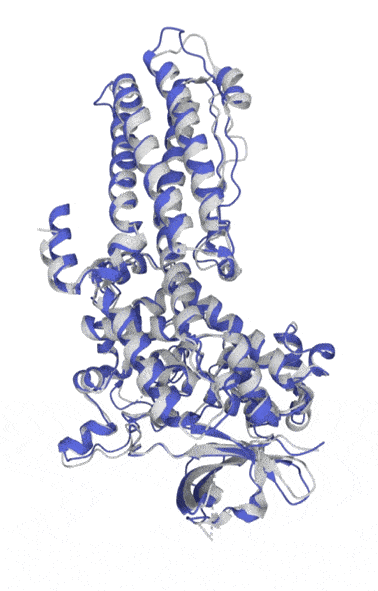
Uni-Fold预测的蛋白结构 。 灰色为实验结果;蓝色为预测结果 。
深势科技方面提到 , 在过去一年里 , 团队克服了算法生态不佳、计算资源有限等难题 , 成功突破技术壁垒 , 复现了AlphaFold2的训练过程 , 并决定将训练代码全部开源 , 让这项高精度的蛋白质结构计算预测软件真正惠及结构生物学领域研究者 。
澎湃新闻采访人员此前报道 , 北京时间7月15日 , DeepMind团队在顶级学术期刊《自然》(Nature)以“加快评审文章”(Accelerated Article Preview)形式在线发表了一篇题为“Highly accurate protein structure prediction with AlphaFold”的论文 , 全面详述了2020年年底造成轰动的这一模型 , 并首次对外分享开源代码 。
DeepMind团队当时向澎湃新闻(www.thepaper.cn)采访人员提供了一份声明 , 公司创始人兼首席执行官Demis Hassabis在声明中表示 , 去年在CASP14大会上我们揭晓了一个可以将蛋白质3D结构预测精确到原子水平的全新AlphaFold系统 , 此后我们承诺会分享我们的方法 , 并为科学共同体提供广泛、免费的获取途径 。
推荐阅读
- 产品|泰晶科技与紫光展锐联合实验室揭牌
- 空间|(科技)科普:詹姆斯·韦布空间望远镜——探索宇宙历史的“深空巨镜”
- 相关|科思科技:无人机地面控制站相关设备产品开始逐步发力
- 机身重量|黑科技眼控对焦23年后回归,升级! 江一白解读EOS R3
- 公司|科思科技:正在加速推进智能无线电基带处理芯片的研发
- 项目|常德市二中2021青少年科技创新大赛再获佳绩
- 视点·观察|科技巨头纷纷发力元宇宙:这是否是所有人的未来?
- 技术|聚光科技旗下临床质谱仪获批医疗器械注册证
- 视点·观察|科技股连年上涨势头难以持续:或已透支未来涨幅
- 视点·观察|科技行业都在谈论“元宇宙”,可是它还不存在