文章图片
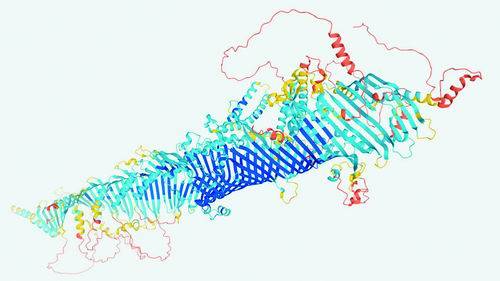
AI现在可以快速可靠地预测大多数蛋白质的三维形状 。 图片来源:DEEPMIND
近日 , 美国华盛顿大学和英国DeepMind公司分别公布了多年工作的成果:先进的建模程序 , 可以预测蛋白质和一些分子复合物的精确三维原子结构 。 其中一个研究小组报告说 , 他们已经使用最新开发的人工智能(AI)程序预测了来自人类和20种模式生物的35万种蛋白质结构——如大肠杆菌、酵母和果蝇 。 未来几个月里 , 他们计划将所有已编目的蛋白质列入模型蛋白质名单 , 大约有1亿个分子 。
“这是相当惊人的 。 ”美国马里兰大学蛋白质专家John Moult说 , 他每两年举办一次名为“关键的蛋白质结构预测方法”(CASP)的比赛 。 Moult表示 , 几十年来 , 结构生物学家一直梦想有一天 , 计算机模型能增加从X射线晶体学等实验方法中获得的极精确蛋白质形状的数量 。 “我从没想过这个梦想会成真 。 ”Moult说 。
这一名为AlphaFold的模型是DeepMind公司研究人员的成果 , DeepMind是一家隶属于谷歌母公司Alphabet的英国AI公司 。 2020年 , AlphaFold“横扫”了CASP 。 但是DeepMind研究人员没有透露绘制蛋白质形状的理论细节 , 特别是AlphaFold的底层计算机代码 。
这种情况已开始改变 。 7月15日 , 华盛顿大学的Minkyung Baek和David Baker研究小组报告说 , 他们创建了一个名为RoseTTAFold的高度精确的蛋白质结构预测程序 , 并公开发布了该程序 。 相关成果在线发表于《科学》 。 与此同时 , 《自然》发表了一篇由DeepMind研究人员Demis Hassabis和John Jumper撰写的论文 , 公布了AlphaFold的细节 。
两个程序都使用AI在庞大的蛋白质结构数据库中识别折叠模式 。 这些程序通过考虑蛋白质中相邻氨基酸相互作用的基本物理和生物学规则 , 计算出未知蛋白质最可能的结构 。 论文显示 , Baek和Baker使用RoseTTAFold创建了一个包含数百个G蛋白偶联受体(一类常见的药物靶点)的结构数据库 。
而DeepMind研究人员创造了35万个预测结构 , 是以前实验方法得到的结果的两倍多 。 研究人员表示 , AlphaFold产生了近44%的人类蛋白质结构 , 涵盖了人类基因组编码的近60%的氨基酸 。 AlphaFold确定许多其他人类蛋白质是“无序的” , 这意味着它们的形状不是单一结构 。
此外 , DeepMind与欧洲分子生物学实验室合作共建了一个新的蛋白质预测数据库 , 可以在网上免费访问 。 “能提供这种服务真是太棒了 。 ” Baker说 , “这真的会加快研究步伐 。 ”因为蛋白质的3D结构在很大程度上决定了它的功能 , 所以这一数据库便于生物学家厘清数千种未知蛋白质是如何工作的 。 (文乐乐)
【梦想成真|梦想成真:AI预测蛋白质结构】[责任编辑: 武玥彤 ]
推荐阅读
- 赛道|声网发布实时互动场景创新生态报告,预测RTE行业市场规模将达千亿
- the|美疾控中心预测:未来一个月内全美或有超4.4万人死于新冠肺炎
- 数字货币|是继续欣欣向荣还是泡沫破灭? 专家给出加密货币2022年五大预测
- the|美国CDC将omicron占比从73%下修 但仍预测病例会继续增加
- 量子|美国《信号》杂志预测2022年十大技术趋势
- 人工智能|人工智能预测蛋白质结构
- 红色星球|外媒预测:40年后,人类太空之旅能走多远
- 预警|200亿美元的智能手表市场又起烽烟:号称能预测心梗、中风,健康预警手表真能救命?
- Tesla|瑞信预测Q4特斯拉交付量同比涨超60% 产量成唯一限制因素
- 中美|2022年十大AI预测:气候独角兽涌现、中美竞争加剧