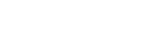混合|拓端tecdat|R语言建立和可视化混合效应模型mixed effect model
原文链接:http://tecdat.cn/?p=20631
我们已经学习了如何处理混合效应模型 。 本文的重点是如何建立和可视化 混合效应模型的结果 。
设置 本文使用数据集 , 用于探索草食动物种群对珊瑚覆盖的影响 。
- knitr::opts_chunk$set(echo = TRUE)
- library(tidyverse) # 数据处理
- library(lme4) # lmer glmer 模型
- me_data <- read_csv("mixede.csv")
。
注意:由于食草动物种群的测量规模存在差异 , 因此我们使用标准化的值 , 否则模型将无法收敛 。 我们还使用了因变量的对数 。 我正在根据这项特定研究对数据进行分组 。
summary(mod)
- ## Linear mixed model fit by maximum likelihood ['lmerMod']
- ##
- ## AIC BIC logLik deviance df.resid
- ## 116.3 125.1 -52.1 104.3 26
- ##
- ## Scaled residuals:
- ## Min 1Q Median 3Q Max
- ## -1.7501 -0.6725 -0.1219 0.6223 1.7882
- ##
- ## Random effects:
- ## Groups Name Variance Std.Dev.
- ## site (Intercept) 0.000 0.000
- ## Residual 1.522 1.234
- ## Number of obs: 32, groups: site, 9
- ##
- ## Fixed effects:
- ## Estimate Std. Error t value
- ## (Intercept) 10.1272 0.2670 37.929
- ## c.urchinden 0.5414 0.2303 2.351
- ## c.fishmass 0.4624 0.4090 1.130
- ## c.maxD 0.3989 0.4286 0.931
- ##
- ## Correlation of Fixed Effects:
- ## (Intr) c.rchn c.fshm
- ## c.urchinden 0.036
- ## c.fishmass -0.193 0.020
- ## c.maxD 0.511 0.491 -0.431
- ## convergence code: 0
- ## boundary (singular) fit: see ?isSingular
plot(mod)
文章图片
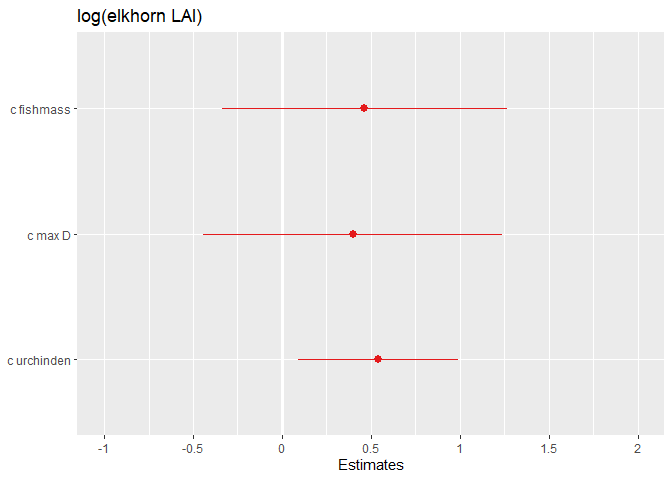
效应大小的格式化图:
让我们更改轴标签和标题 。
- # 注意:轴标签应按从下到上的顺序排列 。
- # 要查看效应大小和p值 , 设置show.values和show.p= TRUE 。 只有当效应大小的值过大时 , 才会显示P值 。
- title="草食动物对珊瑚覆盖的影响")
文章图片
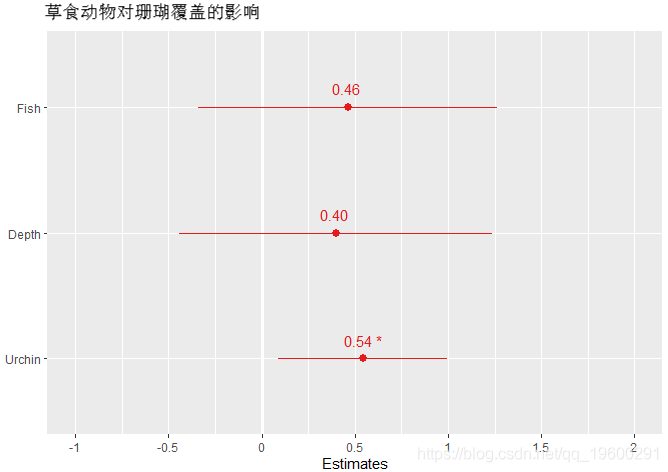
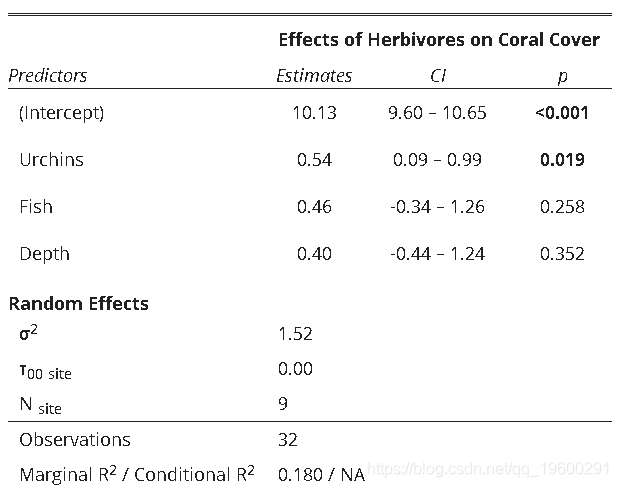
模型结果表输出: 创建模型摘要输出表 。 这将提供预测变量 , 包括其估计值 , 置信区间 , 估计值的p值以及随机效应信息 。
tab(mod)
文章图片
格式化表格
- # 注:预测标签(pred.labs)应从上到下排列;dv.labs位于表格顶部的因变量的名称 。
- pred.labels =c("(Intercept)", "Urchins", "Fish", "Depth"),
文章图片
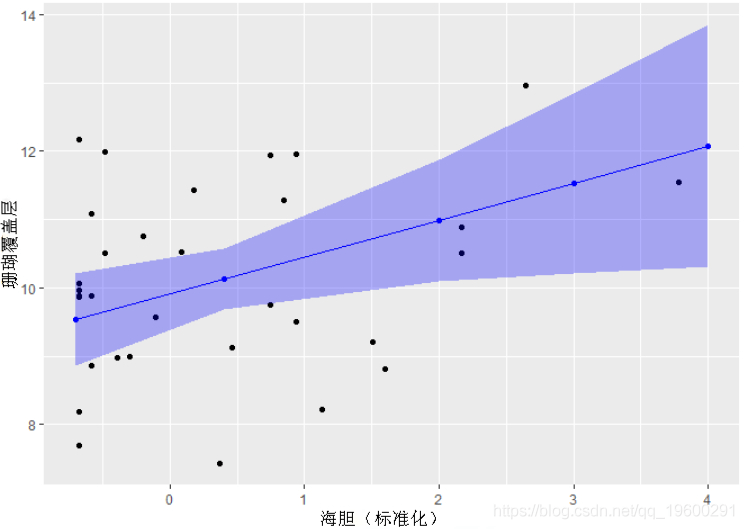
用数据绘制模型估计 我们可以在实际数据上绘制模型估计值!我们一次只针对一个变量执行此操作 。 注意:数据已标准化以便在模型中使用 , 因此我们绘制的是标准化数据值 , 而不是原始数据
步骤1:将效应大小估算值保存到data.frame中
- # 使用函数 。term=固定效应 , mod=你的模型 。
- effect(term= "c.urchinden", mod= mod)
- summary(effects) #值的输出
- ##
- ## c.urchinden effect
- ## c.urchinden
- ## -0.7 0.4 2 3 4
- ## 9.53159 10.12715 10.99342 11.53484 12.07626
- ##
- ## Lower 95 Percent Confidence Limits
- ## c.urchinden
- ## -0.7 0.4 2 3 4
- ## 8.857169 9.680160 10.104459 10.216537 10.306881
- ##
- ## Upper 95 Percent Confidence Limits
- ## c.urchinden
- ## -0.7 0.4 2 3 4
- ## 10.20601 10.57414 11.88238 12.85314 13.84563
- # 将效应值另存为df:
- x <- as.data.frame(effects)
如果要保存基本图(仅固定效应和因变量数据) , 可以将其分解为单独的步骤 。 注意:对于该图 , 我正在基于此特定研究对数据进行分组 。
- #基本步骤:
- #1创建空图
- #2 从数据中添加geom_points()
- #3 为模型估计添加geom_point 。 我们改变颜色 , 使它们与数据区分开来
- #4 为MODEL的估计值添加geom_line 。 改变颜色以配合估计点 。
- #5 添加具有模型估计置信区间的geom_ribbon
- #6 根据需要编辑标签!
- #1
- chin_plot <- ggplot() +
- #2
- geom_point(data , +
- #3
- geom_point(data=https://www.sohu.com/a/x_, aes(x= chinde, y=fit), color="blue") +
- #4
- geom_line(data=https://www.sohu.com/a/x, aes(x= chinde, y=fit), color="blue") +
- #5
- geom_ribbon(data= https://www.sohu.com/a/x , aes(x=c.urchinden, ymin=lower, ymax=upper), alpha= 0.3, fill="blue") +
- #6
- labs(x="海胆(标准化)", y="珊瑚覆盖层")
- chin_plot
文章图片
【混合|拓端tecdat|R语言建立和可视化混合效应模型mixed effect model】

文章图片
最受欢迎的见解
1.基于R语言的lmer混合线性回归模型
2.R语言用Rshiny探索lme4广义线性混合模型(GLMM)和线性混合模型(LMM)
3.R语言线性混合效应模型实战案例
4.R语言线性混合效应模型实战案例2
5.R语言线性混合效应模型实战案例
6.线性混合效应模型Linear Mixed-Effects Models的部分折叠Gibbs采样
7.R语言LME4混合效应模型研究教师的受欢迎程度
8.R语言中基于混合数据抽样(MIDAS)回归的HAR-RV模型预测GDP增长
9.使用SAS , Stata , HLM , R , SPSS和Mplus的分层线性模型HLM
推荐阅读
- 配件|微软配件新品上市:主打混合办公,自带 Teams 认证
- 单元|Astell Kern AK ZERO1 混合单元耳机发售:5299 元,内置平板振膜
- 操作系统|京东云联合信通院发布混合云白皮书,以云原生助力产业数字化
- 业内|爱捷云入选业内首个《混合云产业全景图》
- LG|LG公布新款新空气净化器 将HEPA和UV紫外光杀菌混合
- Apple|苹果混合现实头显Apple View高清概念渲染
- 云建设|爱捷云入选“2021年度混合云优秀案例”
- 数据|混合云:让亚马逊妥协的真相
- DELL|[视频]戴尔构想Stanza/Flow/Pari概念 解决混合办公痛点
- 施工|全球首艘混合动力化学品船“安徽造”