data|拓端tecdat|R语言中生存分析模型与时间依赖性ROC曲线可视化
原文链接:http://tecdat.cn/?p=20650
人们通常使用接收者操作特征曲线(ROC)进行二元结果逻辑回归 。 但是 , 流行病学研究中感兴趣的结果通常是事件发生时间 。 使用随时间变化的时间依赖性ROC可以更全面地描述这种情况下的预测模型 。
时间依赖性ROC定义
令 Mi为用于死亡率预测的基线(时间0)标量标记 。当随时间推移观察到结果时 , 其预测性能取决于评估时间 t 。 直观地说 , 在零时间测量的标记值应该变得不那么相关 。 因此 , ROC测得的预测性能(区分)是时间t的函数。
累积病例
累积病例/动态ROC定义了在时间t 处的阈值c处的 灵敏度和特异性 ,如下所示 。
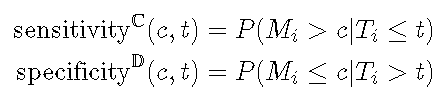
文章图片
累积灵敏度将在时间t之前死亡的视为分母(疾病) , 而将标记值高于 c 的作为真实阳性(疾病阳性) 。 动态特异性将在时间t仍然活着作为分母(健康) , 并将标记值小于或等于 c 的那些作为真实阴性(健康中的阴性) 。 将阈值 c 从最小值更改为最大值会在时间t处显示整个ROC曲线。
新发病例
新发病例ROC1在时间t 处以阈值 c定义灵敏度和特异性 ,如下所示 。
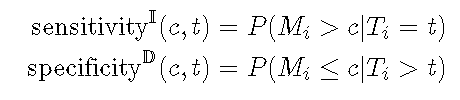
文章图片
累积灵敏度将在时间t处死亡的人 视为分母(疾病) , 而将标记值高于 ? 的人视为真实阳性(疾病阳性) 。
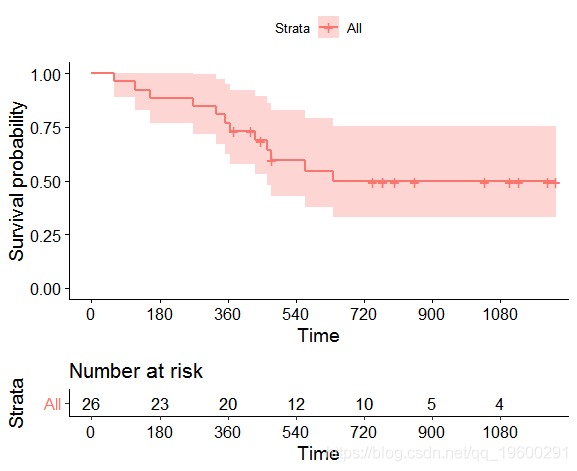
数据准备 我们以数据 包中的dataset3survival为例 。 事件发生的时间就是死亡的时间 。 Kaplan-Meier图如下 。
- ## 变成data_frame
- data <- as_data_frame(data)
- ## 绘图
- plot(survfit(Surv(futime, fustat) ~ 1,
- data = https://www.sohu.com/a/data)
文章图片
在数据集中超过720天没有发生任何事件 。
- ## 拟合cox模型
- coxph(formula = Surv(futime, fustat) ~ pspline(age, df = 4) +
- ##获得线性预测值
- predict(coxph1, type = "lp")
文章图片
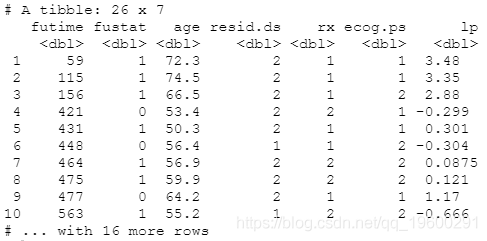
累积病例 实现了累积病例
- ## 定义一个辅助函数 , 以在不同的时间进行评估
- ROC_hlp <- function(t) {
- survivalROC(Stime
- status
- marker
- predict.time = t,
- method = "NNE",
- span = 0.25 * nrow(ovarian)^(-0.20))
- }
- ## 每180天评估一次
- ROC_data <- data_frame(t = 180 * c(1,2,3,4,5,6)) %>%
- mutate(survivalROC = map(t, survivalROC_helper),
- ## 提取AUC
- auc = map_dbl(survivalROC, magrittr::extract2, "AUC"),
- ## 在data_frame中放相关的值
- df_survivalROC = map(survivalROC, function(obj) {
- ## 绘图
- ggplot(mapping = aes(x = FP, y = TP)) +
- geom_point() +
- geom_line() +
- facet_wrap( ~ t) +
文章图片
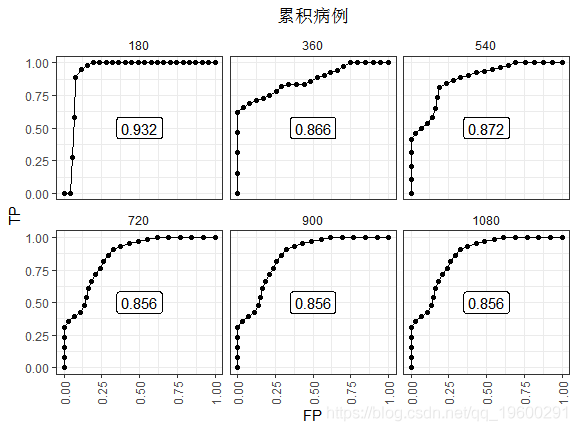
180天的ROC看起来是最好的 。 因为到此刻为止几乎没有事件 。 在最后观察到的事件(t≥720)之后 , AUC稳定在0.856 。 这种表现并没有衰退 , 因为高风险分数的人死了 。
新发病例
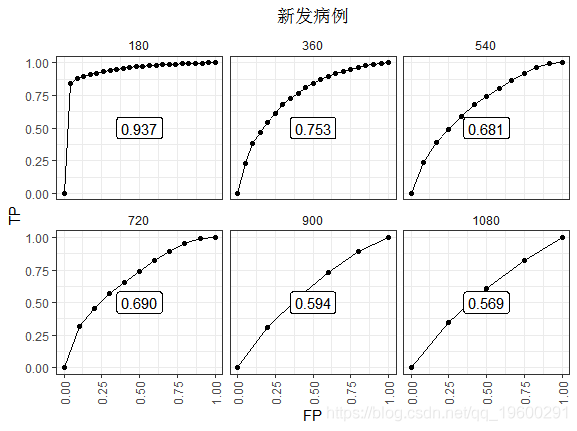
实现新发病例
- ## 定义一个辅助函数 , 以在不同的时间进行评估
- ## 每180天评估一次
- ## 提取AUC
- auc = map_dbl(risksetROC, magrittr::extract2, "AUC"),
- ## 在data_frame中放相关的值
- df_risksetROC = map(risksetROC, function(obj) {
- ## 标记栏
- marker <- c(-Inf, obj[["marker"]], Inf)
- ## 绘图
- ggplot(mapping = aes(x = FP, y = TP)) +
- geom_point() +
- geom_line() +
- geom_label(data = https://www.sohu.com/a/risksetROC_data %>% dplyr::select(t,auc) %>% unique,
- facet_wrap( ~ t) +
【data|拓端tecdat|R语言中生存分析模型与时间依赖性ROC曲线可视化】
文章图片
这种差异在后期更为明显 。 最值得注意的是 , 只有在每个时间点处于风险集中的个体才能提供数据 。 所以数据点少了 。 表现的衰退更为明显 , 也许是因为在那些存活时间足够长的人中 , 时间零点的风险分没有那么重要 。 一旦没有事件 , ROC基本上就会趋于平缓 。
结论 总之 , 我们研究了时间依赖的ROC及其R实现 。 累积病例ROC可能与风险 (累积发生率)预测模型的概念更兼容。 新发病例ROC可用于检查时间零标记在预测后续事件时的相关性 。
参考
- Heagerty,Patrick J. and Zheng,Yingye, Survival Model Predictive Accuracy and ROC Curves,Biometrics , 61(1),92-105(2005). doi:10.1111 / j.0006-341X.2005.030814.x.

文章图片
最受欢迎的见解
1.R语言绘制生存曲线估计|生存分析|如何R作生存曲线图
2.R语言生存分析可视化分析
3.R语言如何在生存分析与Cox回归中计算IDI , NRI指标
4.r语言中使用Bioconductor 分析芯片数据
5.R语言生存分析数据分析可视化案例
6.r语言ggplot2误差棒图快速指南
7.R 语言绘制功能富集泡泡图
8.R语言如何找到患者数据中具有差异的指标?(PLS—DA分析)
9.R语言中的生存分析Survival analysis晚期肺癌患者4例
推荐阅读
- 峰会|TalkingData T11 2021 数据智能峰会线上举办,探寻赋能增长之道
- CBNData|超四成消费者追求家的安全感,家居向全屋智能进阶 |CBNData报告
- 控制|Azure Database for MySQL Flexible Server上线
- 评选|TalkingData入选「爱分析·中国数据智能最佳实践案例」
- 标准|TalkingData获ISO/IEC 27701:2019认证
- Git|JetBrains全新数据科学IDE DataSpell正式发布
- 平台|安全合规的数据合作新模式,TalkingData安全岛助力营销增长
- 参会|虽迟但到,首次线上版TalkingData T11数据智能峰会来了!
- SaaS|清科创业旗下SaaS产品PEDATAMAX荣获「年度数字解决方案奖」
- 硬件|Iodyne推出Pro Data八口雷电SSD存储盒:速度5GB/s 主打工作站应用