机器之心报道
编辑:维度、陈萍
借助深度学习算法 , AI 能够成功地识别出细胞中难以识别的各种不同结构 , 这点人类有时都做不到 。
文章图片
利用人工智能(AI)标记和识别细胞的 3D 结构是一个非常有趣的研究课题 , 需要用到生物学中的荧光显微镜技术和深度学习等 AI 技术 。 艾伦细胞科学研究所(Allen Institute for Cell Science)专注于相关领域的研究 , 并取得了一些成果 。
细胞的 3D 动态影像 。
Susanne Rafelski 是该研究所的定量细胞生物学家和副主任 , 她和她的同事希望为细胞中各种不同的结构打上相应的标签 , 并做成 3D 动态影像 。 不过 , 这个愿望实现起来并不容易 。

文章图片
Susanne Rafelski 。
荧光显微技术(fluorescence microscopy)虽然很常用 , 但在这里遇到了麻烦 。 首先 , 可供使用的颜色很少 , 无法完整地标记细胞结构;其次 , 试剂成本很高且使用起来麻烦;最后 , 染色剂以及成像过程对活细胞有害 。
在这种情况下 , 投射白光(明视野显微镜技术)就派上了用场 , 利用该技术的细胞成像不依赖标记 , 也就不会遇到荧光显微技术带来的一些问题 。
据介绍 , Rafelski 团队将荧光显微技术和投射白光技术结合了起来 , 从而利用人工智能(AI)在明视野图像上预测荧光标记的形状 。 该团队的研究已经持续了数年 。
深度学习发挥重要作用
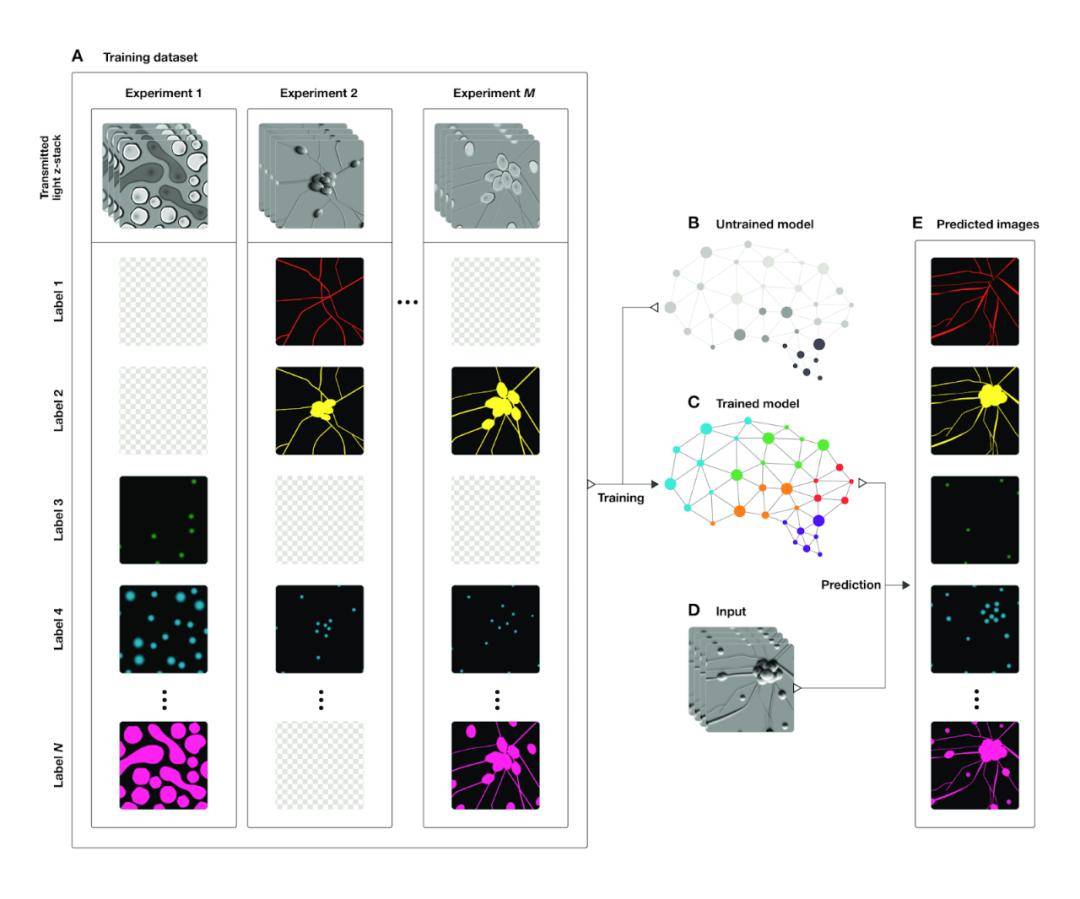
在识别细胞结构的过程中 , 深度学习发挥了重要作用 。 2017 年 , 该团队提出利用深度学习来识别未标记细胞明视野图像中难以发现的结构 , 并证实了这种方法的可行性 。
具体而言 , 通过在未标记细胞实验中使用一种深度学习算法 , 团队创建了一个展示细胞核中 DNA 和子结构、细胞膜和线粒体的 3D 影像 。
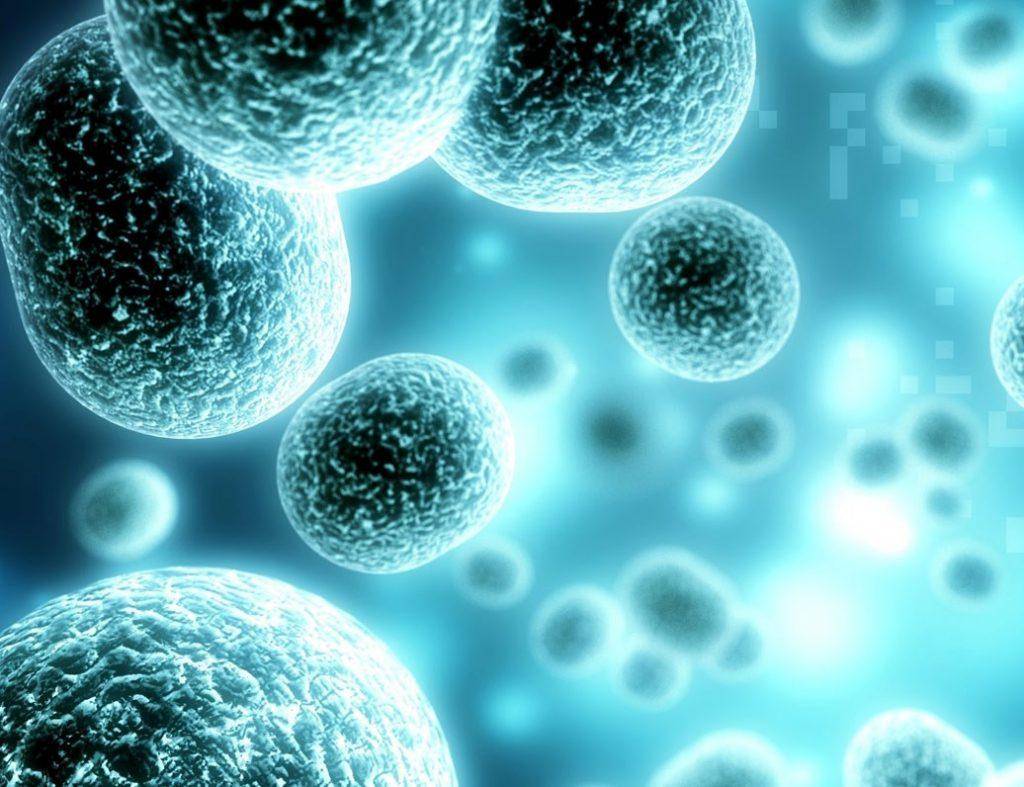
文章图片
基于不同细胞类型的透射光显微镜(明视野)图像输入的 3D 荧光图像预测 。 图源:https://www.nature.com/articles/s41592-018-0111-2#Sec19
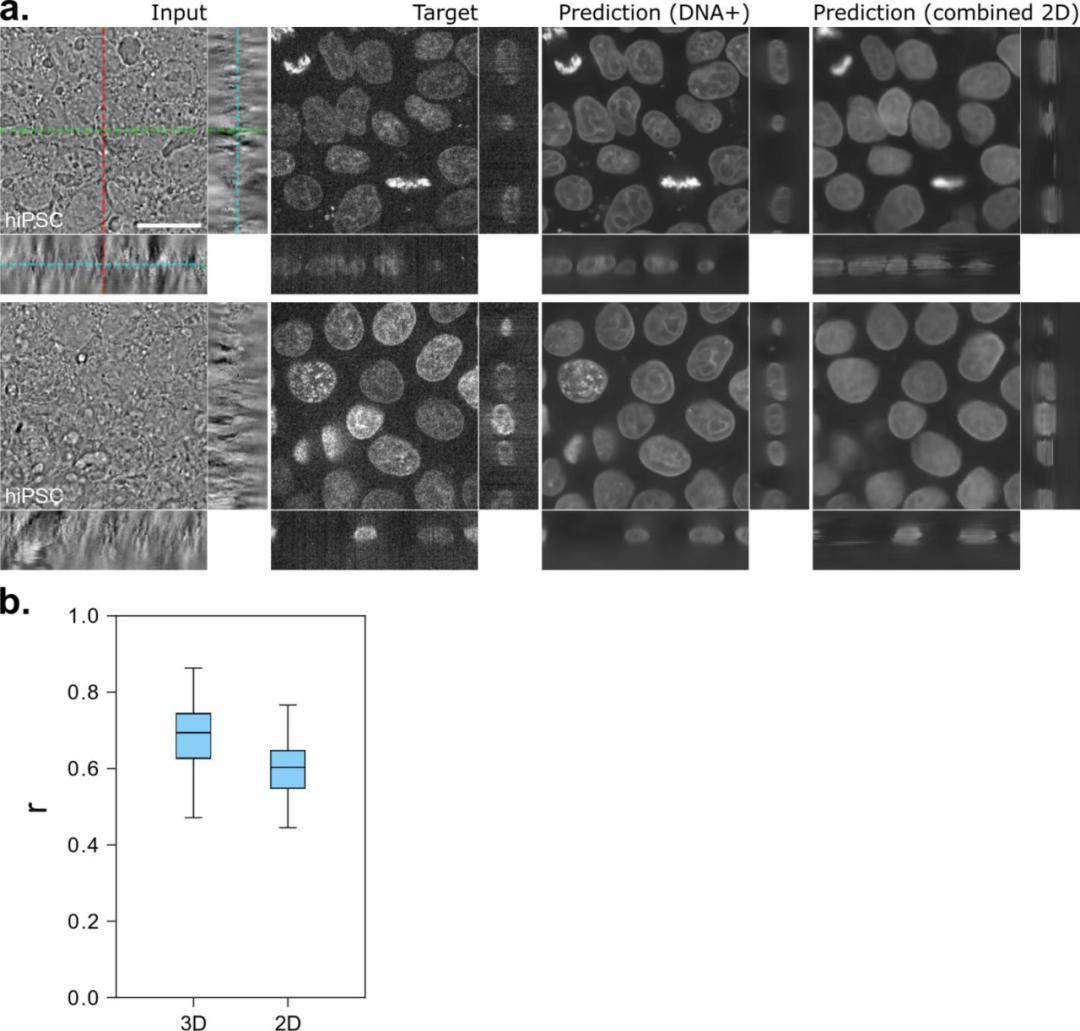
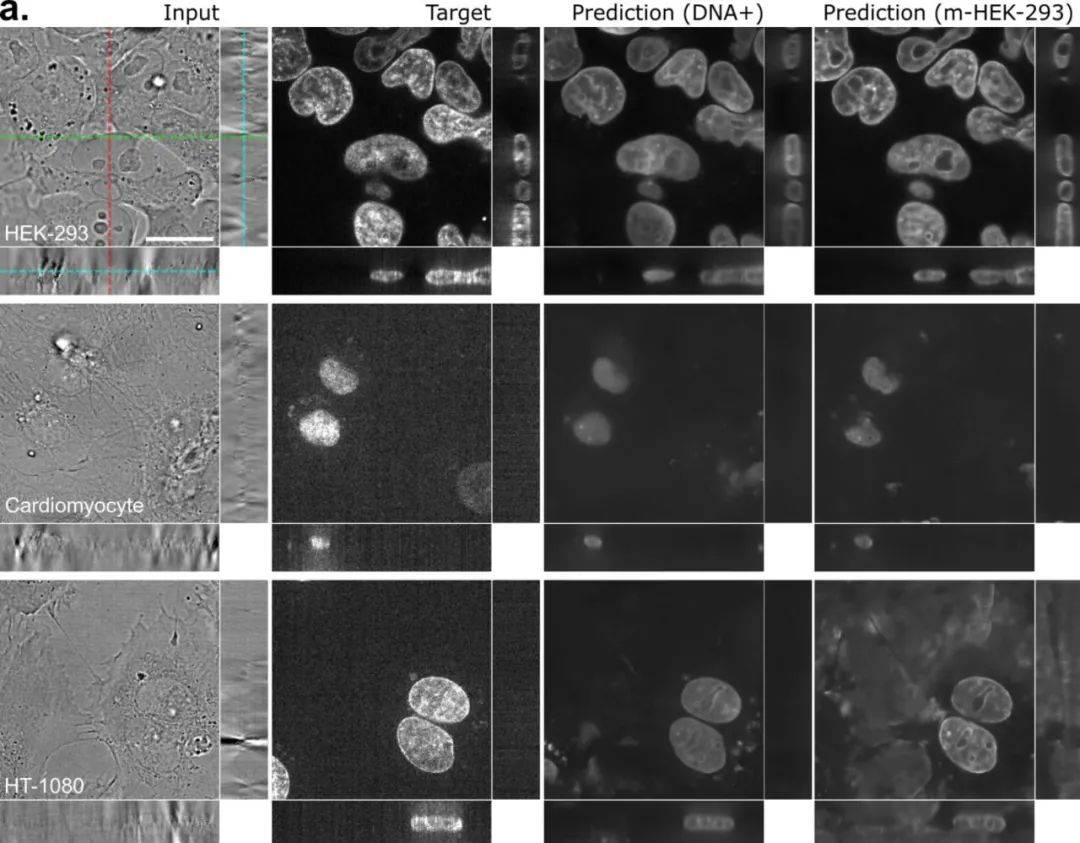
文章图片
分别使用全 3D 和 2D 模型时 , 基于透射光(明视野)图像的 3D DNA 预测 。 图源:https://www.nature.com/articles/s41592-018-0111-2
这种直接从透射图像中预测 3D 荧光的无标记方法可以用于生成多结构、组合式图像 , 也能根据电子显微(EM)输入来预测免疫荧光(IF) , 从而扩展了潜在的应用范围 。
更多研究细节可以参考论文《Label-free prediction of three-dimensional fluorescence images from transmitted-light microscopy》 。
论文链接:https://www.biorxiv.org/content/10.1101/289504v4
AI 识别细胞的发展历程
在过去的几年里 , 从事人工智能研究的科学家们设计了几个系统来识别这些模式 。 使用来自相同细胞的成对图像训练模型、图像分为一个明视野和一个荧光标记 。 但这些模型在细节上有所不同:有些用于 2D 图像、有些用于 3D 图像、有些是用于近似细胞结构 , 而另一些则是用来制作可能被误认为是真实显微照片的图像 。
来自加州大学旧金山分校和旧金山格莱斯顿研究所的神经科学家 Steven Finkbeiner 使用机器人显微镜跟踪细胞长达一年 。 在研究中 Finkbeiner 发现 , 使用深度学习可以发现看不见的细胞特征 。

文章图片
Finkbeiner 团队用训练系统来识别 2D 图像中的神经元 , 然后挑出细胞核 , 确定给定的细胞是否活着 。 他表示说 , 自己研究的主要目的是向科学家表明 , 图像数据中的信息可能比人类意识到的还要多 。 该小组称其技术为「in silico labeling(ISL)」 。 ISL 能直接从未标记的固定样本或活体样本的透射光影像中预测多种荧光标记 。
文章图片
图源:https://ai.googleblog.com/2018/04/seeing-more-with-in-silico-labeling-of.html
然而 , 这种方法无法识别运动神经元 。 这些预测只有在 AI 能够使用一些可见线索的情况下才会起作用 。
Collman、Johnson 以及在艾伦研究所的同事使用了一种不同的神经网络来解决 Rafelski 的问题 , 建立了一个叫做 U-Net 的系统 , 这个系统为生物图像而开发 。 与 Finkbeiner 的方法不同 , Allen 模型可处理 3D 显微照片 , 研究人员可以常规使用该技术 , 例如 , 在染色质组织研究中识别核标记 。

文章图片
透射光显微镜三维荧光图像的无标记预测 。 图源:https://github.com/AllenCellModeling/pytorch_fnet/tree/release_1
来自伊利诺伊大学厄巴纳-尚佩恩分校的物理学家 Gabriel Popescu 正在利用深度学习来回答一个最基本的显微镜问题:细胞是活的还是死的?这是比较难的 , 因为测试需要有毒的化学物质 。 并表示说:这就像用刀测量病人的脉搏 。

文章图片
Gabriel Popescu 。
Popescu 和同事将他们的方法称为 PICS:具有计算特异性的相位成像 。 Popescu 在活细胞中使用该技术来识别细胞核和细胞质 , 然后计算它们在数天内的质量 。 并表明 , 这些信号准确地表明了细胞的生长和生存能力 。
PICS 包含了基于 U-Net 软件和显微镜硬件技术 , 因此 , PICS 不是先获取图像并训练机器 , 之后在进行进一步的处理 , PICS 是无缝地进行 。 一旦用户捕捉到白光图像 , 模型只需 65 毫秒就能传递出预测的荧光对应物 。
除此以外 , 还有其他研究小组使用机器学习来识别细胞 。 例如 , 华盛顿特区美国天主教大学的一个研究小组使用了一种称为 GAN 的神经网络来识别相衬光学显微镜图像中的细胞核 。
参考链接:
https://www.nature.com/articles/s41592-018-0111-2
https://www.nature.com/articles/d41586-021-00812-7#ref-CR1
_原题《在识别细胞结构上 , AI做到了人类不能做的事情》
【识别|在识别细胞结构上,AI做到了人类不能做的事情】阅读原文
推荐阅读
- 星链|石豪:在太空,马斯克和美国当局是如何作恶的
- 建设|这一次,我们用SASE为教育信息化建设保驾护航
- 生活|气笑了,这APP的年度报告是在嘲讽我吧
- 精度|将建模速率提升10倍,消费级3D扫描仪Magic Swift在2021高交会大显“身手”
- 最新消息|世界单体容量最大漂浮式光伏电站在德州并网发电
- 公司|科思科技:正在加速推进智能无线电基带处理芯片的研发
- 测试|图森未来完成全球首次无人驾驶重卡在公开道路的全无人化测试
- 识别|沈阳地铁重大变化!能摘口罩吗?
- 识别|天津滨海机场RFID行李全流程跟踪系统完成建设 行李标签识别成功率可提升至99%
- Monarch|消息称微软Win11 2022重大更新将在明年夏天到来